- 작성일
- 2021.10.21
- 수정일
- 2021.10.21
- 작성자
- 이준희
- 조회수
- 1792
단백질 구조 예측에서의 혁명: 알파폴드와 로제타폴드
출처: [BRIC Bio통신원] [초심자를 위한 생물학+정보학] 단백질 구조 예측에서의 혁명: 알파폴드와 로제타폴드
생명과학 고주온 (2021-10-14)
생체 내외에서 일어나는 많은 현상에는 효소를 비롯한 단백질이 다수 관여한다. 잘 알다시피, 아미노산으로 이루어진 단백질에는 비단 효소 뿐만 아니라 물질 이동과 저장, 구조적 역할, 생리적 기능, 그리고 수용체 등 신호 전달 및 조절 등 다양한 기능을 수행하는 수만 종이 존재한다. 그런데 종류가 다양한 만큼 모양새도 각각 달라서 그 구조를 알아내는 것이 상당히 어렵다. 주어진 1차원의 아미노산 서열만으로 해당 단백질의 3차원 구조를 예측하는 것이 불가능했기 따문에, 현재까지는 주로 X-선 결정학적 방법 (X-ray crystallography), 초저온 전자현미경 (cryo-EM) 등을 이용하여 단백질 실물을 바탕으로 하는 실험을 통해 단백질의 구조를 연구해 왔다. 그러나, 실제 단백질로부터 구조를 알아내는 접근법은 여러 가지 한계를 갖고 있어 아직 그 구조를 알아내지 못한 단백질이 훨씬 많은 것이 현실이다.
단백질의 구조를 파악하는 것이 왜 중요한 지는 사람마다 생각이 다를 수 있다. 그러나 생명 현상 자체가 수많은 반응의 집합체이고 이러한 반응의 주체로서 작용하는 각 단백질의 중요성에는 이론의 여지가 없다. 그리고 단백질이 반응의 대상을 확인하고 이어지는 일련의 과정에서 해당 단백질의 구조적 특성은 매우 중요한 요인으로 작용한다. 따라서 생물학적 반응의 주체로서의 단백질과 그 물리적 분자 구조는 반응의 여러 수준에서 결정적인 영향을 주게 된다.
예컨대 현재까지 어떤 유사한 단백질조차도 구조가 알려져 있지 않은 한 단백질의 기능을 알아 보고자 할 때, 그 구조를 가상의 공간에서 예측할 수 있고 이를 토대로 해당 단백질과 결합 또는 반응하는 물질과의 관계를 모사할 수 있다면, 이는 실제 실험실에서의 실험 설계와 진행에 있어서 노력과 비용, 그리고 신뢰도 등의 여러 측면에서 대단히 중요하고 새로운 진전을 이루게 해 줄 것이다.
단백질 구조 연구 분야에서 세계적으로 유명한 경진대회로 CASP1가 있다. 이 대회는 미국 캘리포니아 대학교 데이비스 캠퍼스 (UC Davis) 소재의 단백질 구조 예측 센터2 주관으로 매 2년마다 열리며, 미 국립 보건원 산하 미 국립 일반 의학 연구소 (NIH/NIGMS)에서 후원한다. CASP는 1994년에 제1회 대회가 시작된 후로, 2년마다 대회를 열어 2020년에는 CASP14가 개최되었다.
이번에 알아 보고자 하는 알파폴드 (AlphaFold)는 2018년 12월 초, 멕시코의 칸쿤 (Riviera Maya, Canc?n, Quintana Roo, Mexico)에서 열린 CASP13에 참가한 구글 (Google)의 자회사 딥마인드 (DeepMind)에서 개발한 도구로서 딥마인드는 참가한 다른 팀에 비해 월등한 성적을 기록하였다. 딥마인드는 2020년 개최된 CASP14에서는 더욱 뛰어난 성능을 발휘하며 다른 참가팀들을 압도하게 되었다 (그림1). 한편, 미국 워싱턴주 시애틀 소재의 워싱턴대학교 (University of Washington, Seattle, WA 98195, USA)의 Baker 연구팀도 CASP13과 CASP14에 모두 참가하였는데, 이들은 CASP14 이후 지난 7월에 로제타폴드 (RoseTTAFold)라는 단백질 구조 예측 도구의 원시코드 (source code)를 오픈소스 (open source)로 공개한 후, 그 내용을 학술잡지 사이언스 (Science)에 출간하였다. 같은 날, 딥마인드도 학술잡지 네이처 (Nature)에 알파폴드에 관한 연구 내용을 발표하면서 동시에 원시코드를 오픈소스로 공개하였다. 이 내용을 일자 별로 간략히 정리하면 다음과 같다.
---
- 2018년 12월 1일 ~ 12월 4일, CASP13 개최 (멕시코 칸쿤).
- 2019년 10월 10일, DeepMind가 CASP13에서 사용한 알파폴드 (AlphaFold)의 알고리듬을 학술잡지 (Proteins, Wiley)에 발표 (Senior et al., 2019).
- 2020년 1월 15일, DeepMind의 AlphaFold에 관한 논문을 Nature에 게재 (Senior et al., 2020).
- 2020년 11월 30일 ~ 12월 4일, CASP14 온라인 개최.
- 2021년 6월, DeepMind가 CASP14 (2020)에서 사용한 알파폴드 2 (AlphaFold v2.0)의 관련 내용을 BioRxiv에 발표3 .
- 2021년 7월 4일, 미국 워싱턴주 시애틀 소재의 워싱턴대학교 (University of Washington, Seattle, WA 98195, USA)의 Baker 연구팀이 개발하여 CASP14 (2020)에서 사용한 로제타폴드 (RoseTTAFold)의 개선판 원시코드를 깃허브 (GitHub)에 공개.
- 2021년 7월 15일, 미국 워싱턴대 Baker 연구팀이 RoseTTAFold 관련 내용을 Science에 발표 (Baek et al., 2021). 같은 날, DeepMind도 알파폴드 2에 관한 논문을 Nature에 게재하고 (Jumper et al., 2021), 원시코드를 GitHub에 공개.
---
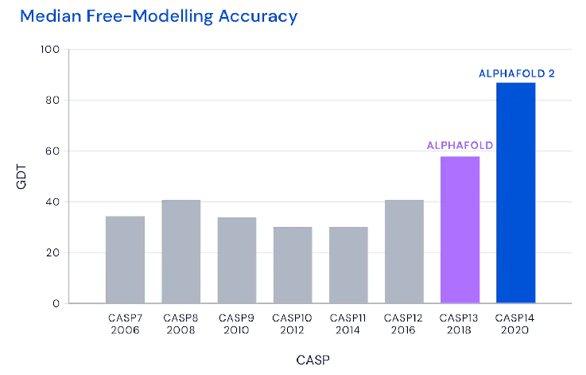
그림 1. CASP의 각 연도별 상위 5개팀에 대한 예측 정확도의 중앙값과 알파폴드의 성적.
(출처: https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology)
2016년 CASP12까지만 해도 우승팀의 단백질 구조 예측 정확도가 50 GDT를 넘지 않고 있었으나, 2018년부터 참가한 딥마인드의 성적이 급격히 올라가다가 2020년의 CASP14에서는 92.4 GDT를 기록함으로써 딥마인드의 알파폴드가 단백질 구조 예측 분야의 새로운 지평을 연 사실만큼은 확실해 보인다. 참고로, CASP13과 CASP14에 참가한 각 팀별 모델 표준 점수와 순위를 보면 극적인 변화를 실감할 수 있다. 특히, CASP14에 들어서 1위팀 (딥마인드)과 2위팀의 격차가 현격하게 벌어진 것을 알 수 있다 (그림 2). 여담이지만, 이를 직접 보고 겪은 미국 워싱턴대학교 Baker 연구팀을 주도한 백민경 박사는 CASP14 당시 어쩌면 곧 직장을 잃게 될 지도 모른다는 생각을 했다고 한다4.
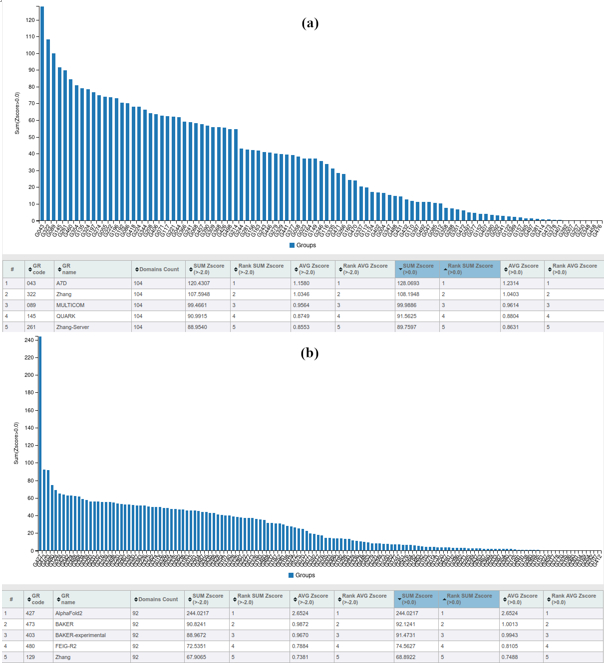
그림 2. CASP13과 CASP14에 참가한 각 팀별 모델 표준 점수 및 순위.
(a) CASP13. (b) CASP14.
그래서인지는 몰라도 2021년에 공개한 로제타폴드의 개선판은 알파폴드 2에 필적하는 성능을 보이는 것으로 평가되고 있다.
로제타폴드는 알파폴드에서 영감을 받아 개발된 것으로 알려져 있으며, 두 도구 모두 심화 학습 (deep learning)의 generative model을 적용하였으나, 그 구체적인 알고리듬과 구현 방법에서 차이가 있다. 또한 이들 도구들은 심화 학습을 구현하기 위하여 막대한 전산 자원을 필요로 하기 때문에 동일한 환경을 개인이 구비하기는 곤란한 측면이 있다. 예컨대, 알파폴드는 12개의 가상CPU (vCPU), 85 Gb의 RAM, 100 Gb의 부트 디스크와 3 Tb의 저장 디스크, 그리고 A100 그래픽 처리 장치 (GPU) 등이 갖춰진 클라우드 환경에서 실행이 검증되었다. 또한, 이미 '알파폴드 단백질 구조 데이터베이스5'가 운영 중에 있고, 각 도구를 이용할 수 있는 웹서비스와 구글의 클라우드를 이용하는 방법6 등의 개발이 빠르게 진행되고 있기도 하다. 그러나, 이들 서비스는 각각이 가지는 한계가 있기 때문에 사용자의 관심 영역에 따라서 직접 도구를 운용할 필요가 생길 수 있다. 이러한 이유로 본 연재에서는 관심이 있는 개인이 시도할 수 있는 최소한의 방법을 도모해 보고자 한다. 따라서, 이 두 도구를 설치하고 사용하는 방법에 대해서 이어지는 연재에서 좀더 자세하게 다루어 보기로 하자.
Baek,M. et al. (2021) Accurate prediction of protein structures and interactions using a three-track neural network. Science, 373, 871?876.
Jumper,J. et al. (2021) Highly accurate protein structure prediction with AlphaFold. Nature, 596, 583?589.
Senior,A.W. et al. (2020) Improved protein structure prediction using potentials from deep learning. Nature, 577, 706?710.
Senior,A.W. et al. (2019) Protein structure prediction using multiple deep neural networks in the 13th Critical Assessment of Protein Structure Prediction (CASP13). Proteins, 87, 1141?1148.
--------------------------------------------
1. the Critical Assessment of techniquies for protein Structure Prediction.
2. Protein Structure Prediction Center (https://predictioncenter.org/).
3. https://www.biorxiv.org/content/10.1101/2021.06.14.448402v1
4. https://www.nature.com/articles/d41586-021-01968-y
5. https://alphafold.ebi.ac.uk/
6. https://colab.research.google.com/github/deepmind/alphafold/blob/main/notebooks/AlphaFold.ipynb
초심자를 위한 생물학+정보학
고주온(필명)
(http://bioprofiler.tistory.com)
IBM-XT시절부터 개인용 컴퓨터를 사용하였으나, 강산이 변한 지금도...
출처: [BRIC Bio통신원] [초심자를 위한 생물학+정보학] 단백질 구조 예측에서의 혁명: 알파폴드와 로제타폴드 - 56 ( https://www.ibric.org/myboard/read.php?Board=news&id=335372 )
- 첨부파일
- 첨부파일이(가) 없습니다.
- 다음글
-
박테리아로 플라스틱 오염 문제 해결한다이준희 2021-11-11 13:53:14.473
- 이전글
-
황현지 한국 미생물학회 최우수 발표상 수상이준희 2021-09-01 17:01:15.59